Comments 22
Из такого способа вычисления номера следует и практический смысл всей затеи. Собственно номера – прикольная штука, но не очень понятно, что с ней дальше делатьЕсли зная номер, можно «нарисовать» дерево, то вроде полезно. Вроде как покомпактнее (занимает меньше места), чем стандартный способ хранения дерева. Привели бы его для сравнения, там вроде ссылки на другие ноды используются?
Не компактнее, по крайней мере для относительно больших деревьев. Стандартный формат записи деревьев в бионформатике примерно такой: (((А, B), (C, D)), E). Буквы — имена листьев, скобки объединяют соседние узлы. Могут быть ещё всякие приблуды для информации о внутренних узлах, но в простейшем случае так. Для данного дерева размер записи 21 символ, считая пробелы. Это, если я ничего не напутал, №8. Три бита. So far, so good. Но размер записи растёт примерно линейно от числа листьев, а номера во всяком случае не медленнее чисел Каталана, то есть сильно хуже, чем квадратично. На практике полный вектор номеров узлов для дерева после десятка листьев весит сильно больше, чем его запись в стандартной нотации.
К тому же такое хранение будет некорректно. С точки зрения номеров ((A, B), C) = (A, (B,C)) = 3, а с точки зрения эволюционной информации в дереве это могут быть сильно разные штуки. Но это я со своей колокольни, конечно. Если кому-то надо хранить десятки тысяч топологий деревьев размером в десятки листьев, то может быть и выгода в хранении.
Их хранят в массивах
Если именно топологию дерева кодировать (без payload в узлах), то достаточно по биту на каждый возможный узел/лист: 2^2^N (Где N — глубина дерева)
Завершающие нули можно отбросить.
Не уверен, что представляю описываемый вами формат хранения. Можете чуть подробнее рассказать или там ссылку кинуть?
Просто в голове засели сбалансированные бинарные деревья
Есть способ хранения бинарных деревьев в массиве:
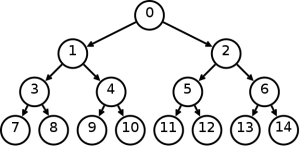
Хранится как:
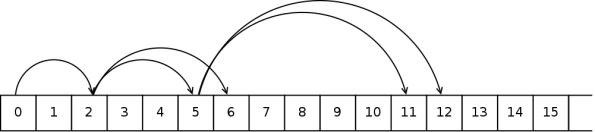
Для отображения только топологии этот массив можно представить в виде бинарного вектора. (1 — есть узел, 0 — нет узла)
Но для разреженных деревьев будет дико неэффективно.
Эффективность будет сильно зависеть от сбалансированности дерева, в особенности количества (k, 1)-поддеревьев. Для каждого из них в правой части создаётся всегда столько же нулей, сколько узлов в k, минус 1. Любые (k,j)-поддеревья будут содержать в правой части меньше нулей, чем (k, 1), и тем меньше, чем k ближе к j. Размер представления получается где-то между 2n-1 и 2^n от числа листьев. Это без удаления завершающих нулей и ещё каких-нибудь оптимизаций.
В общем случае у (несбалансированного) дерева тогда будет несколько представлений
Это в смысле деревья:

и

равны?
Или я чего-то не понял?
Я как раз рассмотрел только простые оптимизации — удаление завершающих нулей и первой единицы.
То есть, если я поменяю в корне правого и левого детей местами, то для вас дерево не изменится?
Я бы сказал максимально минимизирует — достаточно запомнить номер разряда, где кончаются единицы и начинаются нули.
Но в большинстве случаев насколько я знаю, эта информация имеет значение…
Для большинства приложений (индексация данных, например) имеет. Но применительно к конкретно филогенетическим деревьям даже положение корня не всегда имеет значение, то есть деревья ((A, B), (C, D)) и ((C, (A, B)), D) часто считаются эквивалентными. Посмотрите, например, на КДПВ моего прошлого поста, там корня вообще нету. Зато очень важна информация, содержащаяся в листьях.
Больше похоже, что вы под деревом подразумеваете граф
A C
>---<
B DИ дополнительные промежуточные вершины не должны влиять на эквивалентность таких графов.
Скажем, на том же Haskell можно сделать совсем коротко.
import Data.Tree
n = Node "Node"
a = n [n [n [n [n [], n []], n []], n []],
n [n [n [], n []], n [n [], n [n [n [], n []], n []]]]]
b = n [n [n [n [n [], n []], n []], n []],
n [n [n [], n []], n [n [], n [n [], n []]]]]
num :: Tree t -> Int
num (Node _ []) = 1
num (Node _ [t1, t2]) = k * (k - 1) `div` 2 + j + 1
where (k, j) = if n1 > n2 then (n1, n2) else (n2, n1)
(n1, n2) = (num t1, num t2)
Как и можно ожидать, 'num a' возвращает 84, 'num b' возвращает 21.
Для кластеризации вроде как нужно задать метрику между деревьями, то есть определить функцию похожести деревьев, так? На рисунке два похожих дерева, но у одного номер 84, у другого — 21. Ну и как определить расстояние между ними?
А на векторах уже можно определить метрику дистанций (например, евклидову)
Как конкретно метрика определяется, поясните, пожалуйста.
Кажется, что можно найти более адекватные метрики.
Значение обоих инвариантов при заданном количестве узлов n известно для двух предельных деревьев — простой разомкнутой цепочки (максимальное) и для графа-звезды (минимальное). Для первого инварианта D(chain) = n-1, D(star) = 2. Для второго (сумма расстояний): Ds(chain) = (n-1)n(n+1)/6, а для звезды: Ds(star) = (n-1)^2.
Все остальные деревья (тут любые деревья, не только бинарные) расположены между этими предельными дистанциями. То есть по значению данных инвариантов можно судить о близости топологии деревьев — к чему ближе дерево: к цепочке (растянутое) или звезде (компактное). Во всяком случае метрика на основе расстояний выглядит прозрачной и обоснованной.
Нумерация двоичных деревьев