Эксперимент по секвенированию транскриптома (RNA-seq) стал практически рутинной процедурой для изучения как модельных организмов, так и для сельскохозяйственных культур. В результате биоинформатической обработки таких экспериментов получаются объемные разнородные данные, представленные нуклеотидными последовательностями транскриптов, аминокислотными последовательностями и их структурно-функциональной аннотацией. Полученные данные важно представить широкому кругу исследователей в виде баз данных (БД). В публикации мы рассмотрим гибридный подход к созданию молекулярно-генетических баз данных, которые содержат информацию о последовательностях транскриптов и их структурнофункциональной аннотации. Сущность подхода в одновременном хранении в БД информации как структурированного типа, так и слабо структурированных данных. Технология использована для реализации БД транскриптомов сельскохозяйственных растений. В публикации рассматриваются особенности реализации такого подхода и примеры формирования как простых, так и сложных запросов к такой базе данных на языке SQL. Данная статья является укороченным пересказом нашей работы doi: 10.17537/2020.15.455 в которой я являюсь соавтором.
Введение
Изучение транскриптомов растений с помощью высокопроизводительного секвенирования (секвенирование РНК, RNA-seq) широко используется в настоящее время для решения таких задач как оценка экспрессии генов для разных генотипов и в разных условиях среды, идентификация последовательностей РНК (для не модельных организмов), поиск маркеров к функционально важным генам. Эксперимент RNA-seq стал практически рутинной процедурой для изучения как модельных организмов (Arabidopsis thaliana), так и для сельскохозяйственных культур (томат, кукуруза, ячмень, пшеница и др.). Результаты транскриптомного эксперимента представляют собой короткие фрагменты нуклеотидных последовательностей и лишь биоинформатическая обработка, включающая несколько стадий, позволяет получить на их основе последовательности транскриптов и их функциональную аннотацию. Именно результаты биоинформатической обработки представляют интерес для биолога и могут быть интерпретированы в терминах функций генов, их продуктов, уровней экспрессии, генетических вариаций и т.п. Необходимо отметить, что в результате транскриптомного эксперимента получается большое количество данных (десятки и сотни тысяч последовательностей), свободный и удобный доступ к которым важно предоставить широкому кругу биологов, далеких от рутинной биоинформатической обработки результатов секвенирования. Этой цели служат базы данных, имеющие удобный пользовательский интерфейс и организующие связи между биологическими последовательностями и их функциональной аннотацией. Среди таких баз можно указать Expression Atlas Европейского института биоинформатики, EGENES, базу данных информации о метаболических путях генов, основанную на транскриптомных данных, базы данных по экспрессии генов для определенных видов организмов: TodoFirgene для пихты Abies sachalinensis; атлас экспрессии генов для розы; базу аннотированных транскриптомов приморской сосны EuroPineDB и др.
Результаты обработки RNA-seq экспериментов, представленные в таких базах данных, являются комплексными и включают: последовательности транскриптов, их локализацию в геноме, классификацию по типам генов (мРНК, днкРНК, миРНК, тРНК и пр.), функциональную аннотацию транскриптов, оценку уровней экспрессии, оценку вариантов изоформ транскриптов. Эти результаты представлены в виде бинарных и текстовых файлов в различных форматах. Это могут быть файлы последовательностей (форматы FASTA, FASTQ), выравниваний (форматы BAM, SAM, PSL и т.д.) или разметки (BED, GFF, GTF). Результаты анализа дифференциальной экспрессии генов обычно представляют в виде таблиц (форматы TSV, XLS). Подобные хорошо структурированные данные удобно описывать в виде классической реляционной модели отношений RDBMS (Реляционная система управления базами данных, англ. Relational DataBase Management System): например, у одного гена может быть несколько изоформ, в эксперименте рассматриваются несколько образцов ткани разных различных особей и т.д.
Отметим, однако, что наряду с хорошо структурированными данными, в результате анализа RNA-seq экспериментов генерируются слабо структурированные и неструктурированные данные, которые не могут быть описаны с помощью реляционной модели. Сложности могут возникать в силу разнородности данных, получаемых в процессе выполнения биоинформатических вычислительных конвейеров. Эта разнородность обусловлена разнообразием методов, которые вовлечены в конвейеры биоинформатической обработки транскриптомных данных. Узлами в конвейерах являются различные программы, которые реализуют методы обработки данных. На практике обычно бывает так, что в существующий вычислительный конвейер для решения какой-то задачи в процессе работы вносятся изменения: например, происходит замена некоторых узлов на новые, которые реализуют более точные или более производительные методы обработки данных. Поэтому заранее невозможно полностью декларировать структуру данных, которая будет получена в результате их обработки. Описание таких слабоструктурированных данных удобнее делать с использованием технологий NoSQL (не только SQL, англ. not only SQL).
В настоящей работе мы предлагаем комплексный подход к описанию транскриптомных данных, который заключается в использовании элементов RDBMS при описании хорошо структурированных данных и NoSQL для описания слабо структурированных данных, полученных в результате широкомасштабного биоинформатического анализа транскриптомных экспериментов у пяти сельскохозяйственных растений (кукурузы, риса, ячменя, томата и картофеля). Анализ этих данных был направлен на идентификацию новых транскриптов, которые либо не выравниваются на референсный геном растения, либо выравниваются на его неаннотированные участки и, таким образом, представляют собой новую, ранее неисследованную часть транскриптома. На примере задачи массового анализа транскриптомов сельскохозяйственных растений мы предлагаем наборы реляционных отношений для описания основных сущностей: исследование, эксперимент, нуклеотидные и белковые последовательности. В то же время, для каждой из этих сущностей мы предлагаем вводить возможность для аннотации наборами слабоструктурированных данных, формат представления которых может быть заранее неизвестен. На основе предложенного гибридного подхода разработана база данных OORT (Out Of Reference Transcripts), которая позволяет пользователям, с помощью поисковых запросов, извлекать информацию о структуре и функциях ранее неаннотированной части транскриптомов сельскохозяйственных растений, в частности: идентифицировать новые гены устойчивости к заболеваниям и абиотическому стрессу, длинные некодирующие РНК, последовательности мРНК, получать оценки уровня экспрессии этих транскриптов. База данных построена на основе анализа 1241 транскриптомных экспериментов и содержит информацию о 20440228 нуклеотидных и 4055996 аминокислотных аннотированных последовательностях. Функциональные возможности базы данных OORT демонстрируются на примере нескольких запросов.
Содержание БД
Содержание БД OORT включает:
метаинформацию о библиотеках транскриптомных экспериментов;
нуклеотидные последовательности транскриптов, полученные в результате de novo сборки;
аминокислотные последовательности, полученные в результате трансляции нуклеотидных последовательностей транскриптов, кодирующих белки;
аннотации нуклеотидных последовательностей (предсказание кодирующего потенциала, выравнивание на референсный геном, оценку уровня экспрессии);
аннотации аминокислотной последовательности (предсказание функциональных доменов, ассоциированные с последовательностью термины онтологии генов).
При работе с базой данных для пользователя важно решать ряд поисковых задач, связанных с идентификацией последовательностей в базе данных по метаинформации об эксперименте, принадлежности к организму, гомологии, функциональным характеристикам, уровню экспрессии транскрипта. Подобный поиск может осуществляться как для нуклеотидных, так и для аминокислотных последовательностей. При этом следует принимать во внимание отношения между аминокислотной последовательностью и нуклеотидной последовательностью транскрипта, из которого она была получена.
Формирование базы данных, индексация
В качестве СУБД при формировании базы данных OORT мы использовали PostgreSQL версии 12. С ее помощью была построена реляционная схема данных связывающая исследования, эксперименты, контиги и белки (см. раздел “Структура базы данных”). База данных позволяет определять реляционные отношения с помощью первичных и внешних ключей.
С помощью языка программирования Python данные из файлов c результатами биоинформатической обработки (см. раздел “Содержание БД OORT”) были преобразованы в структуру типа “словарь”, которую затем конвертировали в формат JSON с помощью библиотеки SQLAlchemy. SQLAlchemy позволяет транслировать код на языке Python в команды на языке SQL и передавать их на выполнение СУБД, получая результаты запросов в виде объектов языка Python. Далее эти данные были преобразованы в структуры формата JSONB для дальнейшего хранения и доступа.
Создаваемая нами схема базы данных разрабатывалась для поиска информации, представленных как в типизированных (хорошо структурированных), так и в слабоструктурированных полях. Типизированные поля предназначены для описания сущностей в БД, типы которых известны и определены однозначно. Например, последовательность представлена в виде текстовой строки, длина последовательности описана полем типа int, название организма описано в виде текстового поля. Для поиска информации в типизированных данных (int, real, и text) проводилась индексация с помощью структуры B-tree, которая в дальнейшем позволяла выполнять, в том числе, и операции поиска с учетом отношений “равно”, “больше”, “меньше”. Структура B-tree позволяет оптимизировать выполнение запросов с указанными операциями.
Помимо типизированных, хорошо структурированных данных, содержимое базы включало слабо структурированную информацию. К таким данным относились аннотации аминокислотных последовательностей в терминах онтологии генов (GO) и белковых доменов; результаты выравнивания контигов на референсные геномы; результаты оценки уровня экспрессии транскриптов и т.д. Данные подобного сорта представлялись в виде текстовых строк в формате JSON: записи вида ключ-значение без определенных заранее ограничений. СУБД PostgreSQL 12 позволяет хранить информацию в формате JSON в структуре реляционной базы как поля специального типа JSONB. Это данные в формате JSON, представленные в бинарном виде для быстрого обращения без повторного синтаксического разбора, при этом ключи и значения в этом поле не описываются в формате языка SQL. Поверх ключей поля JSONB можно создавать индексы для быстрого поиска данных, также некоторые индексы позволяют использовать новые поисковые опции над значениями, например, поиск искомого объекта в массиве. Индексация полей формата JSON проводилась с помощью GIN индексов. Такая индексация позволяет быстро производить поиск текстовых строк в указанном массиве терминов, например, терминов онтологии и белковых доменов.
Для индексации координат контигов в геноме использовалась структура “материализованное представление”, специальная техника, реализованная в СУБД PostgreQSL, которая позволяет сохранить в виде отдельной таблицы базы данных результат определенного запроса [41]. Это позволяет, после выполнения запроса в виде “материализованного представления”, обращаться к нему как к отдельной таблице реляционной БД. Такая технология позволяет существенно ускорить выполнение запросов (особенно сложных) к базе данных, поскольку в случае повторного запроса обращение производится только к этой таблице, а не к реальным полям БД.
Доступ к информации в БД
Доступ к извлечению и модификации данных в БД OORT производился на языке запросов SQL. Типичный запрос на поиск данных включает, как правило, следующие секции этого языка:
SELECT– секция выбора полей таблиц. Здесь через запятую нужно указать имена колонок, которые нужно отобразить, или * чтобы отобразить все поля.FROM– секция таблиц. В этой секции следует указать таблицы, из которых нужно получить результаты. Также в этой секции может использоваться оператор JOIN, с помощью которого выполняется объединение таблиц.WHERE– секция условия. В ней пользователь указывает условия поиска и объединяет их с помощью логических операторов AND, OR и NOT (условие не выполняется).
Следует отметить, что СУБД PostgreSQL версии 12 расширяет диалект языка SQL:2016 за счет дополнительных операторов, которые служат для извлечения данных из полей формата JSONB. К этим операторам относятся оператор '->', который используется для извлечения вложенного JSON-значения по ключу, и оператор '->>', который используется для получения строковых значений по ключу. Использование этих операторов продемонстрировано ниже в разделе “Примеры поисковых запросов”. Интерфейс SQL к разработанной БД OORT может быть реализован с помощью таких приложений, как DataGrip, pgAdmin или консольного приложения psql СУБД PostgreSQL.
Структура базы данных
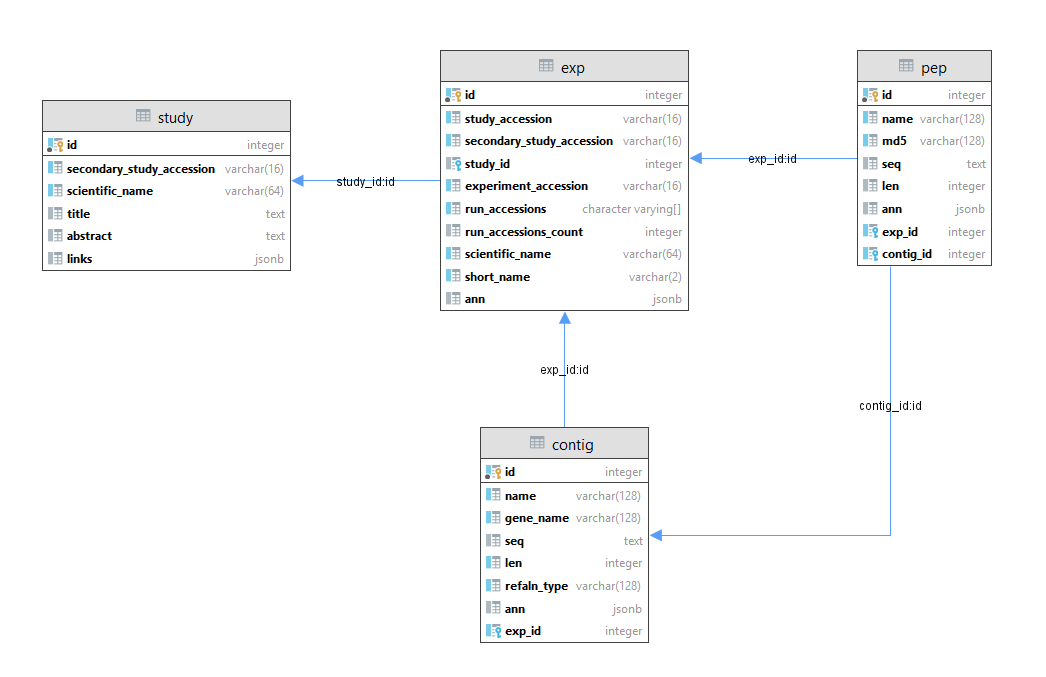
Реляционная схема базы данных включает описание метаданных транскриптомного эксперимента и результатов сборки транскриптов de novo в виде следующих объектов: исследование (study), эксперимент (exp), нуклеотидная последовательность (contig) и аминокислотная последовательность (pep) (рис. 1). Как было указано в разделе “Формирование базы данных, индексация”, при создании ссылок между таблицами БД мы использовали первичные и вторичные ключи, которые были созданы следующим образом:
каждая из таблиц БД хранит поле id, которое является первичным ключом;
таблица exp содержит вторичный ключ study_id, который ссылается на таблицу study;
таблица contig содержит вторичный ключ exp_id, который ссылается на таблицу exp;
таблица pep содержит вторичные ключи contig_id и exp_id, которые ссылаются на таблицы contig и exp, соответственно.
В результате, между указанными таблицами были созданы реляционные связи, позволяющие эффективно осуществлять поиск информации по эксперименту, исследованию и полученных в результате эксперимента последовательностях. Структура отношений между таблицами БД OORT представлена на рисунке 1.
Количество данных в БД OORT представлен в таблице. Общий объем данных составил 45.5 гигабайт.
Название таблицы | Количество записей | Размер данных |
Описание исследований | 2766 | 3.0 МБ |
Описание экспериментов | 1241 | 7.4 МБ |
Нуклеотидные последовательности | 20440228 | 39000.0 МБ |
Аминокислотные последовательности | 4055996 | 6495.0 МБ |
Пример описания слабоструктурированных данных
Рассмотрим подробнее способ описания слабо структурированных данных в БД OORT. Эти данные в указанных выше таблицах ‘exp’, ‘pep’ и ‘contig’ реализованы в виде текстовых полей типа jsonb, которые содержат результаты аннотации последовательностей. Рассмотрим пример описания результатов оценки уровня экспрессии транскрипта TRINITY_DN3631_c0_g1_i1 программой kallisto в формате JSON в поле ‘ann’ таблицы ‘contig’ БД OORT:
"kallisto": {
"g": {
"tpm": 8.44,
"eff_length": 341.61,
"est_counts": 35.0
},
"i": {
"tpm": 8.43596,
"eff_length": 341.614,
"est_counts": 35.0
}
}Тут представлена часть содержимого поля ‘ann’ для таблицы ‘contig’ транскрипта TRINITY_DN3631_c0_g1_i1, которая описывает оценку уровня экспрессии, полученную в результате использования программы kallisto. Эту информацию в поле ‘ann’ можно выделить по названию ключа “kallisto” (первая строка записи). Программа kallisto дает оценку уровня экспрессии как для гена, к которому относится транскрипт (раздел записи, который соответствует ключу “g”, вторая строка), так и для собственно транскрипта, представляющего одну из изоформ данного гена (раздел записи, который соответствует ключу “i”, седьмая строка). Для каждой такой оценки уровня экспрессии программа kallisto выдает три параметра: “tpm”, оценка уровня экспрессии в величинах "транскрипт на миллион”, TPM; “eff_length”, эффективная длина последовательности; “est_counts”, оценка числа прочтений, выравненных на данный транскрипт. Все эти параметры приводятся в соответствующих значениях для ключей, указанных в формате JSON.
Аналогичным образом описывается аннотация аминокислотных последовательностей. Описание результатов аннотации белковых доменов программой Interproscan содержит разделы:
tsv – двумерный массив вывода программы InterproScan;
go_ann – массив строк с GO аннотациями белка;
pathways_ann – массив строк со списком сигнальных и метаболических путей, в которых участвует белок.
Описание результатов предсказания открытой рамки считывания и ее параметров программой TransDecoder содержит разделы:
type – строка с обозначением типа предсказанной рамки трансляции (полная, от старт до стоп-кодона, либо ее фрагмент);
chain – строка с обозначением направления цепи, с которой происходит трансляция (“+” или “–”);
score – число, характеризующее надежность идентификации открытой рамки трансляции в нуклеотидной последовательности;
cds_end – число: координата конца открытой рамки трансляции в нуклеотидной последовательности;
cds_start – число: координата начала открытой рамки трансляции.
Примеры реализации поисковых запросов к БД
Приведем примеры ряда запросов на языке СУБД PostgreSQL 12 к БД OORT. Первый запрос заключается в получении всех последовательностей транскриптов, полученных из библиотек риса (O. sativa). Данный запрос для выполнения использует только стандартные операторы SQL обращения к полям БД:
-- Запрос 1: Получить последовательности генов для вида 'Oryza sativa'
select contig.seq
from contig join exp on exp.id = contig.exp_id
where scientific_name = 'Oryza sativa'Второй запрос заключается в получении списка идентификаторов всех генов, уровень экспрессии которого находится в пределах от 3 до 7 TPM. Этот запрос использует обращение к полю JSON ‘ann’ и извлекает из него значение по ключу “kallisto” с использованием операторов -> и ->>.
-- Запрос 2: Найти все гены, с уровнем экспрессии в пределах от 3.0 до 7.0 TPM
select *
from contig
where ((ann -> 'kallisto' -> 'g' ->> 'tpm')::real)
between 3.0 and 7.0Третий запрос заключается в поиске генов, аминокислотные последовательности которых идентичны (т.е. имеют одинаковые значения хэш-кода md5), при этом уровень их экспрессии в разных экспериментах различается.
-- Запрос 3: Вывести список генов, кодирующих идентичные
-- пептиды с разным уровнем экспрессии в разных экспериментах
select subseq.exp_id, subseq.study_id,
max(subseq.contig_g_tpm) as contig_g_tpm,
study.scientific_name, tissue_type
from (
select (contig.ann->'kallisto'->'g'->'tpm')::real
as contig_g_tpm, contig.id as contig_id, pep.id as
pep_id, contig.exp_id, e.study_id, pep.md5,
e.ann->'ebi'->'tissue_type' as tissue_type
from pep join contig on pep.contig_id = contig.id
join exp e on contig.exp_id = e.id
where pep.md5 = '857c1634328c5333ab73efcc11a45038'
)
subseq join study on subseq.study_id = study.id
group by exp_id, study_id, study.scientific_name,
tissue_typeЗапрос 4 заключается в поиске аминокислотных последовательностей, которые в своей аннотации содержат определенные термины онтологии генов, предсказанные программой InterproScan. Так как информация об аннотации не типизирована, то перед выполнением этого запроса необходимо произвести GIN индексацию поля ‘ann’ таблицы ‘pep’ для значений ключа “go_ann”. Это позволяет искать записи уже в индексном массиве.
-- Запрос 4: Найти все аминокислотные последовательности
-- с определенным набором терминов генной онтологии
-- cоздание индекса для выполнения запроса
create index ix_pep_ann_interpro_go_ann on pep using
gin (((ann -> 'interpro'::text) -> 'go_ann'::text))
-- поиск белков согласно аннотации генной онтологии на основе созданного индекса
select * from pep
where (ann -> 'interpro' -> 'go_ann')
?& array['GO:0055085', 'GO:0006811']Запрос 5 заключается в поиске всех исследований, представленных в БД OORT, которые в описании содержат термин ‘Categorizing’. Для его реализации на первом этапе создается GIN индекс на вектора слов из поля ‘abstract’ (4 строка, метод to_tsvector), где записаны краткие описания исследования. Далее выполняется запрос к этим векторам (строки 9–10).
-- Запрос 5: Поиск слова в описаниях исследования
-- создание индекса для полнотекстового поиска
create index study_abstract_idx on study
(to_tsvector('english'::regconfig, abstract));
-- выполнение полнотекстового поиска
select *
from study
where to_tsvector(study.abstract)
@@ plainto_tsquery('Categorizing')Запрос 6 заключается в поиске белков, которые синтезируются с транскриптов, выравненных на участок генома с заданными координатами программой gmap. Так как ключ “gmap” имеет структуру двумерного массива, то на первом этапе эти массивы нормализуются в “материальном отображении” таким образом, чтобы в одной строке таблицы была одна строка из исходного массива (строки 2–9). Далее индексируются три позиции в полученной таблице (строки 12–15) и выполняется поиск искомых последовательностей (строки 19–30).
-- реализация материального представления contig_gmap_view_sqlalchemy
create materialized view
contig_gmap_view_sqlalchemy as
select db.id, jsonb_array_elements(
db.ann -> 'gmap'::text) AS gmap
from (
select contig.id, contig.ann
from contig ORDER BY contig.id
) db;
-- создание индексов поверх колонок GMAP
create index gmap_start_end_chr_idx
on contig_gmap_view_sqlalchemy
(((gmap ->> 15)::integer), ((gmap ->> 16)::integer),
(gmap ->> 13));
-- поиск белков, синтезирующихся со 2 хромосомы с 2000 по 5000
-- позиции в геноме
select pep.id AS pep_id
from exp join contig ON exp.id = contig.exp_id
join contig_gmap_view_sqlalchemy
on contig.id = contig_gmap_view_sqlalchemy.id
join pep ON pep.contig_id = contig.id
where
cast(contig_gmap_view_sqlalchemy.gmap ->> 15
as integer) >=2000 AND
cast(contig_gmap_view_sqlalchemy.gmap ->> 16
as integer) <= 5000
and(contig_gmap_view_sqlalchemy.gmap ->> 13) = '2'
and exp.scientific_name like 'Zea mays'Приведенные примеры показывают, что к существующей БД можно организовывать достаточно сложные запросы, которые могут включать как информацию о последовательности, ее локализации в геноме, аннотации. Запросы могут учитывать результаты сравнения различных характеристик последовательностей, описанных в БД.
Созданная нами база данных на основе гибридного метода описания комплексных молекулярно-биологических данных в виде, как хорошо структурированных (типизированных) данных, так и слабо структурированной информации, обладает рядом преимуществ. Прежде всего, такая организация хранения данных облегчает их сопровождение: при изменении набора программ аннотации, их версий или набора выводимых ими параметров, вместо того, чтобы модифицировать реляционную схему базы и повторно загружать данные в базу, разработчик может составлять слабосвязанные структуры в формате JSON.
Предложенная архитектура БД сохраняет связность таблиц при помощи методов первичных и вторичных ключей. Следует отметить, что структура JSON-описания нетипизированных данных строго не определена и для разных записей даже одной таблицы может отличаться. В конечном итоге, эта структура зависит от того, какие данные были внесены в конкретное поле БД. Поддержание однородности данных в формате JSON при такой организации лежит на разработчике: контроль структуры полей JSON происходит либо в момент экспорта данных, и\или путем реализации функций-обработчиков, которые запускаются при каждом обновлении и добавлении данных.
Выводы
В работе предложен гибридный подход к созданию баз молекулярно-генетических данных, которые содержат информацию о последовательностях транскриптов и их структурно-функциональной аннотации. Сущность подхода заключается в одновременном хранении в БД информации как структурированного типа, так и слабо структурированных данных. Данная схема обладает рядом преимуществ в сопровождении данных: вместо того, чтобы модифицировать реляционную схему базы и повторно загружать данные в базу, разработчик может составлять слабосвязанные структуры в формате JSON, и также производить индексацию поля внутри этой структуры.
Цитирование
Вы можете использовать материалы данной публикации в любых целях. При этом, пожалуйста, цитируйте первоисточник doi: 10.17537/2020.15.455.